Сегодня генетическая модификация растений, особенно сельскохозяйственных культур, — широко распространенная практика. Ее используют для изменения характеристик плодов, повышения урожайности и устойчивости растений к стрессовым воздействиям. Очень важную роль в процессе модификации играет технология CRISPR/Cas.
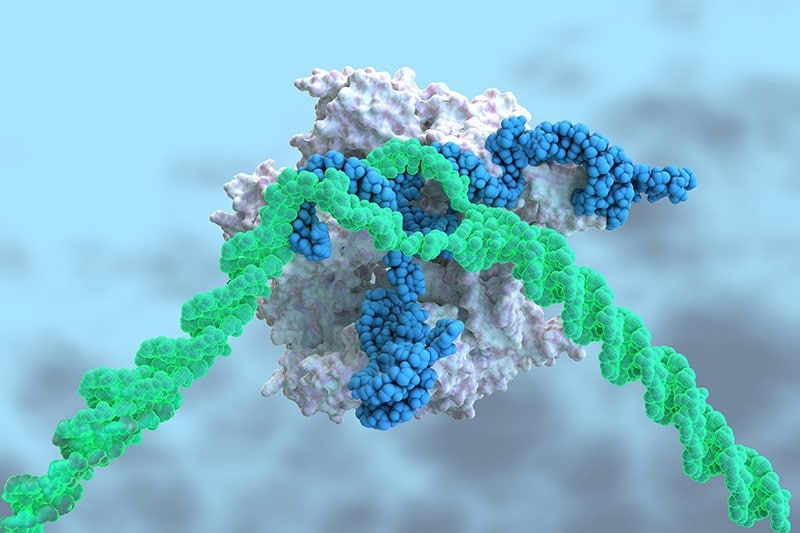
В современной генной инженерии CRISPR/Cas — «рабочая лошадка» большинства методов направленного редактирования генов. При помощи нее можно целенаправленно вырезать участки ДНК в клетках различных организмов, чтобы на их место встроить другие участки ДНК, несущие нужный ген. Целевой участок ДНК опознается направляющей (или гидовой) РНК (гРНК), после чего белок Cas его вырезает.
В теории все звучит просто, однако на практике есть риск неспецифического редактирования ДНК (не только в том месте, где предполагалось) и нарушения последовательности кодирующих генов. Все из-за того, что гРНК может быть комплементарна нескольким участкам ДНК. Ситуация усугубляется в случае многих сельскохозяйственных культур из-за сложности и полиплоидности их геномов.
Решением задачи адаптации технологии CRISPR/Cas для геномов растений решили заняться в Центре передовых инноваций в области биоэнергетики и биопродуктов (CABBI). Исследователи разработали CROPSR — первый в своем роде программный инструмент с открытым исходным кодом для полногеномного проектирования и оценки последовательностей гРНК для экспериментов CRISPR/Cas. Результаты работы опубликованы в журнале BMC Bioinformatics.
«CROPSR предоставляет научному сообществу новые методы и новый рабочий способ проведения экспериментов с нокаутом CRISPR/Cas9, — поясняет разработчик CROPSR, ведущий автор исследования и аспирант по молекулярной биологии Ханс Мюллер Пауль (Hans Müller Paul). — Мы надеемся, что новое программное обеспечение ускорит проведение исследований и снизит количество неудачных экспериментов».
Дело в том, что существующие программные инструменты, необходимые для разработки и оценки эффективности экспериментов CRISPR, до сих пор основывались на редактировании геномов бактерий и млекопитающих, которые очевидно отличаются от полиплоидных геномов сельскохозяйственных культур с кратным набором хромосом. В растениях один признак — например, связанный со стрессом, где полезны резервные системы — может регулироваться набором генов.
Ученый может разработать эксперимент с CRISPR/Cas-системой, чтобы отключить один или несколько генов и не знать о других, выполняющих ту же функцию. Он также может не знать о существовании похожей последовательности ДНК другого гена с иной функцией, который может быть случайно вырезан.
В любом случае проблему можно не выявить до тех пор, пока растение не вырастет и не созреет, без какого-либо изменения признака или с какой-нибудь новой характеристикой (очень редко полезной). Эта проблема особенно остро проявляется с культурами, для выращивания которых требуются особые погодные условия, когда пропуск сезона может означать задержку разработки на год.
Для решения этой задачи команда создала программное обеспечение (автономный пакет, написанный на Python), в котором пересмотрела подход к разработке и оценке последовательностей гРНК с учетом того, что необходимо вырезать все копии гена и не задеть ненужные участки ДНК. По словам авторов, модели оценки гРНК в CROPSR обеспечивают куда более точные прогнозы применения CRISPR/Cas даже в геномах, не относящихся к сельскохозяйственным культурам.
Разработчики также заложили в CROPSR возможность создания базы данных гРНК для всего генома сельскохозяйственной культуры. Этот процесс требует значительных вычислительных ресурсов и времени.
Но исследователям нужно сделать это всего один раз, чтобы создать базу данных, которую затем можно неоднократно использовать для многих экспериментов.
В результате все, что нужно сделать ученому, — найти нужный ген в собственной базе данных, выбрать гРНК из предлагаемого списка, а CROPSR сам укажет на другие места в геноме, на которые нужно нацелиться.
«Вы можете просто зайти в базу данных, получить всю необходимую информацию, готовую к работе, и начать исследование, — заключает Пауль. — Чем меньше времени вы тратите на планирование своих экспериментов, тем больше времени можете потратить на сами эксперименты».